Research
My research activities are concentrated around computational anatomy and image processing applied to medical imaging, ranging from segmentation of soft structures to voxel comparison of different imaging modalities and follow-up sessions at the population level, e.g. detecting differences due to a specific pathology or treatment.
Keywords: Computational anatomy, Magnetic resonance imaging, Image segmentation, Image registration, Neuromuscular disorders, Pelvic floor dynamics
Students
read_more
Supervision of Salomé Baup - 2024 - Master project in Life Sciences Engineering - École Polytechnique Fédérale de Lausanne
Subject: Deep learning-based automatic segmentation of the left ventricle in innovative 5D free-running cardiac magnetic resonance images
Subject: Deep learning-based automatic segmentation of the left ventricle in innovative 5D free-running cardiac magnetic resonance images
Abstract
In this project, the regularization weights for compressed-sensing (CS) reconstruction of free-running (FR) cardiac magnetic resonance (MR) images are optimized for gradient echo sequence at 3 Tesla, preserving both cardiac motion and image quality. Deep-learning-based automatic segmentation of the reconstructed FR images is performed achieving reliable segmentation of the left ventricular cavity (LVC) and left ventricular myocardium (LVM) with results comparable to state-of-the-art methods and variability similar to inter-expert divergences. 5-fold cross validation is performed on a dataset of 20 subject with 10 healthy volunteers, 5 patients with heart failure with preserved ejection fraction and 5 patients with heart failure with reduced ejection fraction. Obtained DSC for the LVC are 0.954 ± 0.012 and 0.915 ± 0.032 in end-diastole and end-systole respectively, and 0.860 ± 0.020 and 0.890 ± 0.013 for the LVM in end-diastole and end-systole respectively.
read_more
Co-supervision of Marc-Adrien Hostin - 2020 - Master 2 internship, Grenoble INP - Phelma
Subject: Segmentation d’IRM pelvienne par apprentissage profond
Subject: Segmentation d’IRM pelvienne par apprentissage profond
Abstract
Les problèmes pelviens touchent près de la moitié des femmes de plus de 50 ans, et peuvent devenir très gênants, jusqu’à nécessiter une opération chirurgicale dans certains cas. Pour reconnaître ces problèmes, les médecins analysent en routine clinique des images IRM 2D dynamiques du plancher pelvien, mais remarquer et localiser les irrégularités sur celles-ci peut être complexe. Pour faciliter l’observation il faudrait pouvoir observer les déformations des organes mous en 3D au cours du temps. L’acquisition IRM en 3D est une opération longue incompatible avec l’observation en temps réel. Pour atteindre le résultat la stratégie proposée par le projet DynPelvis3D consiste à faire des observations partielles mais rapides dans des plans 2D distincts. Pour permettre sa reconstruction en 3D l’objet observé sur ces plans doit être segmenté afin d’extraire la localisation des organes des images IRMs acquises. Le travail de ce stage porte sur l’étape de segmentation car celle-ci peut s’avérer très longue et laborieuse, et les méthodes de segmentations automatiques traditionnelles ne donnent pas de résultats assez convaincants sur le plancher pelvien. On s’intéresse donc ici à l’utilisation de l’apprentissage profond pour la segmentation des organes pelviens, et en particulier au réseau de neurone convolutif U-Net pour la segmentation de la vessie, en cherchant notamment à maximiser les résultats en variant les paramètres du réseau et en développant de nouvelles méthodes liées à l’apprentissage profond.
read_more
Co-supervision of Marouane El Amiri - 2019 - Master 1 internship, CENTRALE Marseille
Subject: Utilisation du deep learning dans la segmentation d’images IRM
Subject: Utilisation du deep learning dans la segmentation d’images IRM
Abstract
Les problèmes pelviens touchent près de la moitié des femmes de plus de 50 ans, et peuvent devenir très gênants, jusqu'à nécessiter une opération chirurgicale dans certains cas. Pour reconnaître ces problèmes, les médecins analysent souvent des images IRM 2D dynamiques du plancher pelvien, mais remarquer et localiser les irrégularités sur celles-ci peut être complexe. C'est pourquoi l'équipe dans laquelle j'ai effectué mon stage travaille sur la reconstruction 3D dynamique des organes pelviens à partir d'images en 2D. Cependant il faut préalablement extraire la localisation des organes des images IRMs acquises : cette étape s'appelle la segmentation. Celle-ci peut s'avérer très longue et laborieuse, et les méthodes de segmentations automatiques traditionnelles ne donnent pas de résultats assez convaincants sur le plancher pelvien. On s'intéresse donc ici à l'utilisation du deep learning pour la segmentation des organes pelviens, et en particulier au réseau de neurone convolutif U-Net pour la segmentation de la vessie, en cherchant notamment à maximiser les résultats en variant les paramètres du réseau.
read_more
Co-supervision of Armando Garcia Hernandez - 2018 - Master 2 internship, Aix-Marseille Université
Subject: Quantitative assessment of pelvic organ deformation
Subject: Quantitative assessment of pelvic organ deformation
Abstract
Pelvic floor disorders affect an important number of women. They are characterized by a debilitating mechanical stability of pelvic organs. This brings symptoms that disturb immensely the patient’s quality of life. Available treatment options range from drug therapy, to implants and in some cases surgical repair. To maximize the success of surgery and reduce the probability of recurrence a precise planning based on Medical Imaging is required. Traditional techniques such as Fluoroscopy, Ultrasound and Magnetic Resonance Imaging are normally used for qualitative assessment of pelvic floor disorders. The aim of this project is to perform a quantitative assessment of pelvic organ deformation using a shape descriptor based on image moments. A shape descriptor based on Zernike moments was implemented and tested. Simulation and in vivo experiments were performed on the bladder to validate its performance.
read_more
Co-supervision of Marine Poutet - 2017 - Master 1 internship, ECE Paris
Subject: Propagation de la segmentation individuelle des muscles d’une IRM de la cuisse au cours du suivi d’un sujet
Subject: Propagation de la segmentation individuelle des muscles d’une IRM de la cuisse au cours du suivi d’un sujet
Abstract
La segmentation manuelle et automatisée des muscles individuels dans les images par résonnance magnétique a été reconnue comme difficile étant donné la discontinuité ou l’absence de limites visibles entre les muscles. Dans ce présent rapport de stage, nous avons proposé un algorithme qui, à partir d’une segmentation réalisée sur une image d’un sujet suite à une première acquisition, propose la segmentation automatique des muscles individuels des images de ce même sujet à d’autres acquisitions. Grâce à des points caractéristiques extraits automatiquement et mis en correspondance entre ces deux images, nous avons pu montrer que le guidage du recalage non-linéaire améliore la propagation de la segmentation, avec un coefficient de similarité Dice supérieur à 0,94.
Projects
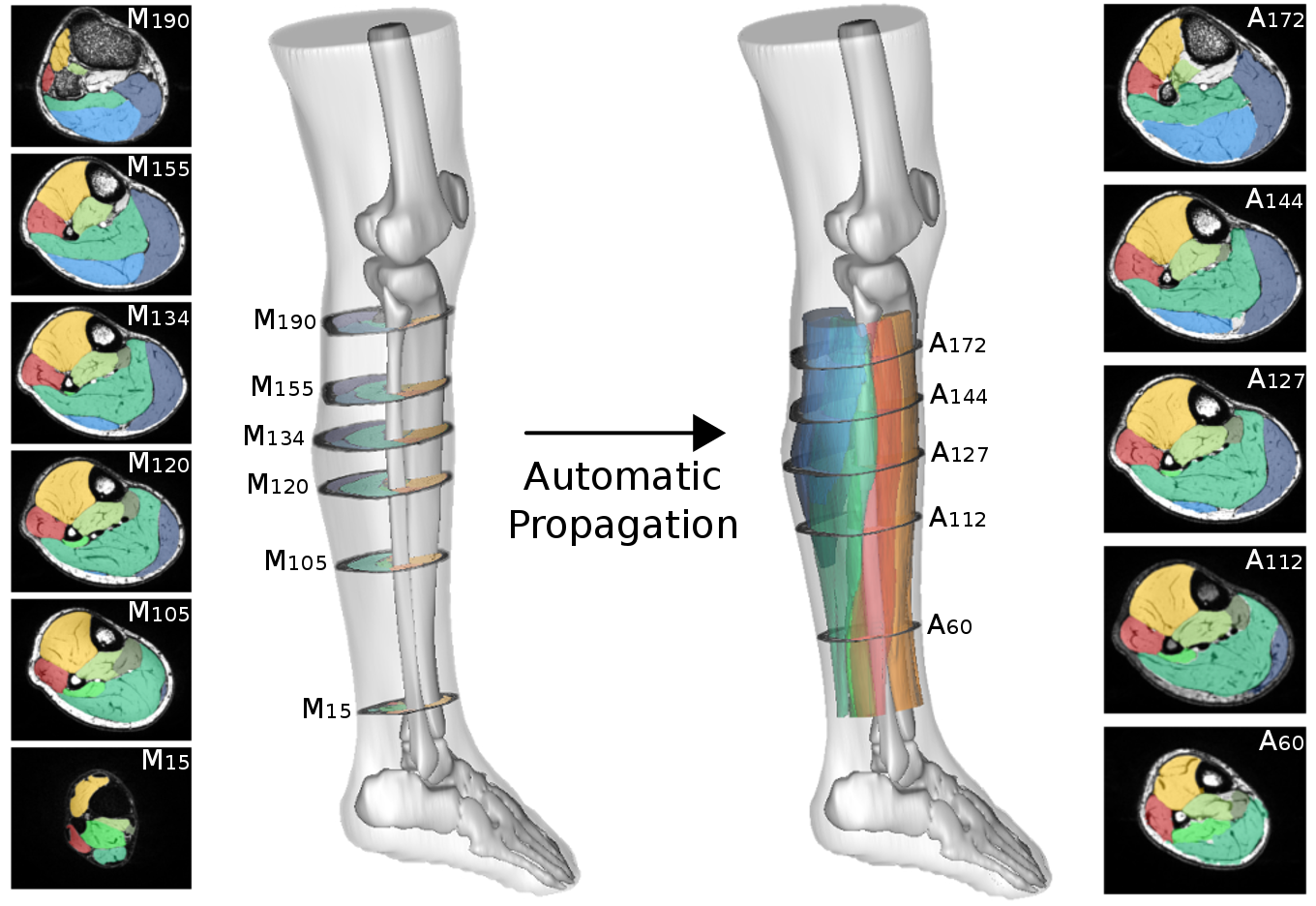
Semi-automatic segmentation by propagation
Computational anatomy aims to develop computer processing methods to enrich diagnosis by extracting objective and clinically useful information from medical images. The deployment of such methods remains limited for the exploration of soft tissue organs, especially for the study of pelvic floor disorders and neuromuscular diseases via magnetic resonance imaging. In these domains, the segmentation step is essential to allow the characterization of physiological alterations occurring within the organs of interest. The high phenotypic variability in these pathologies has so far limited the development of robust automatic segmentation methods, limiting clinical research on large populations. To overcome this challenge, the main contribution of my thesis was the development of a supervised segmentation approach based on diffeomorphic image registration propagation methods.
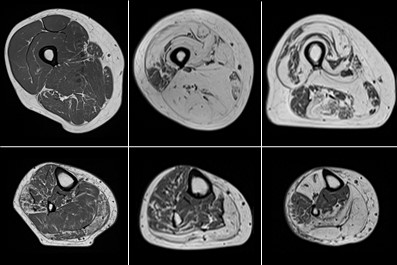
Characterization of physiological alterations in skeletal muscle by quantitative MRI
The development of segmentation methods for muscle soft tissues has allowed the 3D exploration of MRI images on large populations of patients with neuromuscular diseases. The transposition of our methodology into user-friendly tools has enabled us to overcome the barrier of clinical use. Therefore, in collaboration with clinicians from the neurology department of the CHU de la Timone in Marseille, several studies have been conducted on the characterization of physiological alterations occurring at the level of individual muscles in various pathologies and during physical exercises. These studies have notably confirmed the potential of quantitative MRI scores as biomarkers of neuromuscular pathologies.
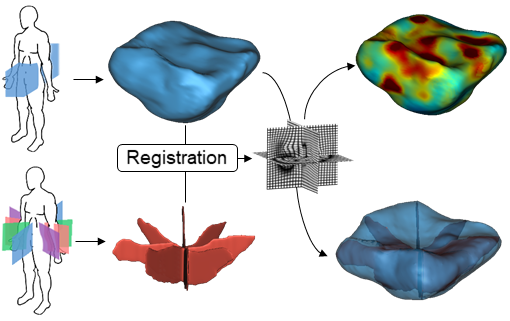
3D reconstruction and characterization of pelvic organ deformations
We proposed the development of a comprehensive methodology for 3D+t representation of pelvic organs. We combined state-of-the-art multi-planar 2D dynamic MRI techniques with our semi-automated segmentation method and geometric non-linear registration approaches to reconstruct dynamic 3D volumes of pelvic organs during loading exercises. To our knowledge, our approach has been the first to propose a dynamic 3D observation of the pelvic region as well as a dynamic 3D representation of the organs. Moreover, the organ reconstruction process allowed a direct characterization of the 3D deformations undergone by the pelvic organs during loading exercises.
